| ที่มา | มติชนสุดสัปดาห์ ฉบับวันที่ 21 - 27 กรกฎาคม 2566 |
|---|---|
| คอลัมน์ | Biology Beyond Nature |
| ผู้เขียน | ภาคภูมิ ทรัพย์สุนทร |
| เผยแพร่ |
ดีเอ็นเอหนึ่งกรัมบรรจุข้อมูลได้กว่า 200 เพนตะไบต์ (200 ล้านจิกะไบต์)
ภายใต้สภาวะเหมาะสมข้อมูลในดีเอ็นเอคงสภาพอยู่ได้เป็นล้านปีโดยแทบไม่ต้องใช้พลังงานเพื่อดูแลรักษา
ด้วยคุณสมบัติสุดพิเศษนี้ทำให้หลายทีมวิจัยทั่วโลกสนใจจะใช้ดีเอ็นเอเป็นตัวกลางบรรจุข้อมูล (data storage medium) อะไรก็ได้ที่เราสนใจ ไม่จำกัดเพียงแค่ “ข้อมูลพันธุกรรม” ตามฟังก์ชั่นธรรมชาติของมันเท่านั้น
แม้แต่บริษัทชั้นนำอย่างไมโครซอฟต์และไอบีเอ็มก็ลงทุนทำวิจัยจริงจังกับเทคโนโลยีด้านนี้
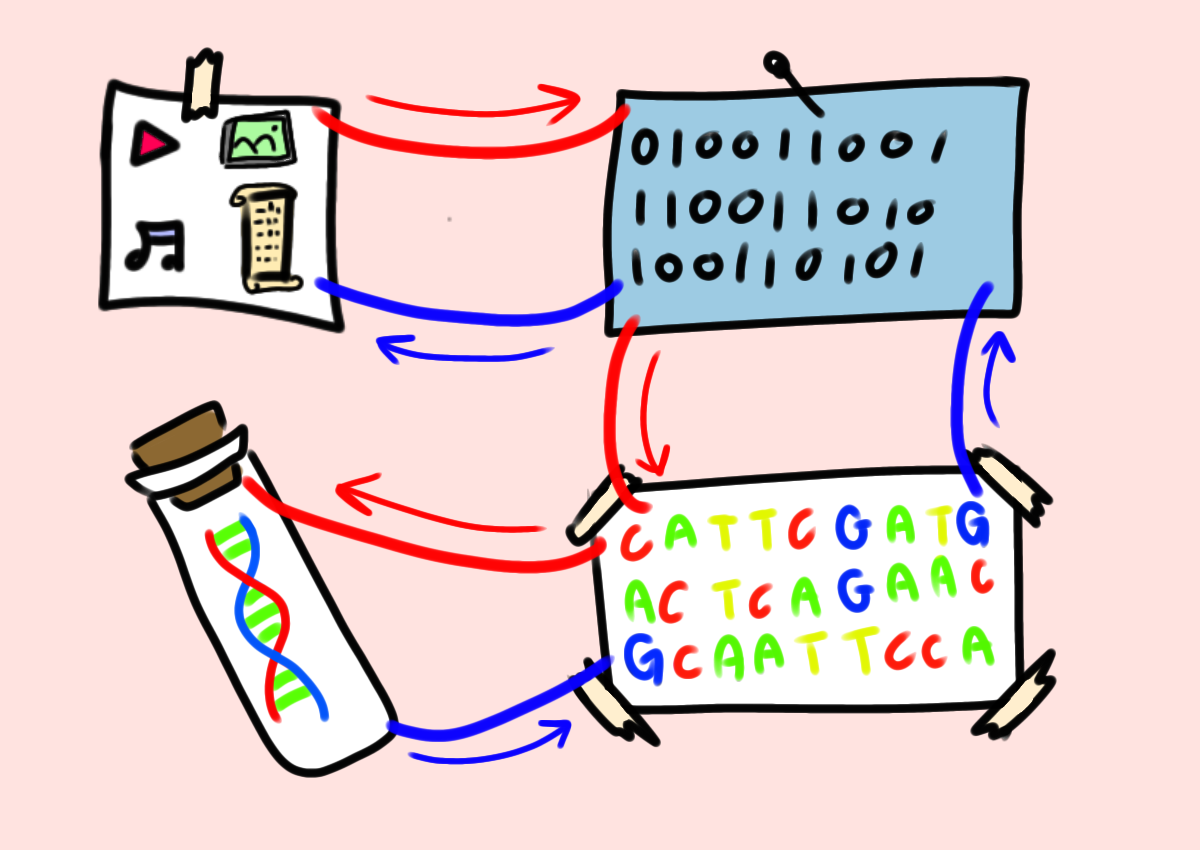
ช่วงไม่กี่สิบปีที่ผ่านมาเทคโนโลยีเก็บข้อมูลลงดีเอ็นเอก้าวกระโดดจากการบรรจุข้อมูลเล็กๆ อย่างรูปโลโก้ขนาดไม่ถึงสิบไบต์มาจนถึงการบรรจุรูป เพลง วิดีโอ หนังสือ ซอฟต์แวร์ ฯลฯ ขนาดกว่าร้อยเมกะไบต์
การบันทึกข้อมูลลงดีเอ็นเอที่ผ่านแทบทั้งหมดใช้ดีเอ็นเอที่สังเคราะห์จากกระบวนการเคมี ไฟล์ข้อมูลดิจิทัลที่ต้องการบันทึกต้องถูกแปลงเป็นเลขฐานสี่ที่สามารถแทนด้วยลำดับเบส A, T, G, C ของดีเอ็นเอ (แทนที่จะเป็นเลขฐานสองที่แทนด้วย 0, 1 อย่างไฟล์ดิจิทัลปกติ)
ข้อมูลลำดับเบสจากไฟล์ถูกใช้สังเคราะโมเลกุลดีเอ็นเอ
ดีเอ็นเอสังเคราะห์อาจจะถูกเก็บเป็นผงแห้งๆ ไว้หรือไม่ก็ฝากใส่เซลล์สิ่งมีชีวิต พอถึงเวลาใช้ก็อ่านลำดับเบสกลับมา (DNA sequencing) แล้วแปลงกลับเป็นไฟล์ตั้งต้นใหม่
วิธีการดังกล่าวทำให้เราบันทึกไฟล์อะไรก็ได้ลงดีเอ็นเอก็จริง
แต่ก็ยังติดปัญหาเรื่องความแม่นยำและราคาของการสังเคราะห์ดีเอ็นเอด้วยกระบวนการทางเคมี
ที่สำคัญกว่านั้นคือการแปลงข้อมูลดิจิทัลลงดีเอ็นเอยังต้องอาศัยมนุษย์ดำเนินการ
ดังนั้น วิธีนี้ทำได้เฉพาะในห้องปฏิบัติการที่มีเครื่องไม้เครื่องมือและนักวิจัยผู้ชำนาญพร้อมเท่านั้น
ในทางกลับกันเซลล์สิ่งมีชีวิตตามธรรมชาติมีกลไกสังเคราะห์และปรับแก้ดีเอ็นเอที่แม่นยำ (และฟรี) อยู่แล้ว
จะดีกว่าไหมถ้าเราจะวิศวกรรมเซลล์ให้สามารถบันทึกข้อมูลอะไรก็ได้ที่เราสนใจลงดีเอ็นเอแบบอัตโนมัติ?
เซลล์ที่บันทึกข้อมูลลงดีเอ็นเออัตโนมัติแบบนี้ต้องมีระบบเซ็นเซอร์ทำหน้าที่ “รับสัญญาณข้อมูล” ที่เราสนใจ
และระบบเซ็นเซอร์นี้ก็ต้องเชื่อมต่อกับอีกระบบที่ทำหน้าที่ “บันทึกข้อมูล”
ลงดีเอ็นเอด้วยการไปปรับแก้ลำดับเบสเป็นสัญลักษณ์สอดคล้องกับข้อมูลนั้นๆ ระบบเซ็นเซอร์ในเซลล์ตามธรรมชาติส่วนมากใช้รับสัญญาณเคมี (ระดับความเข้มข้นของโมเลกุลต่างๆ รอบเซลล์)
ดังนั้น งานวิจัยแนวนี้แทบทั้งหมดจะเกี่ยวกับการบันทึกข้อมูลสัญญาณเคมีลงดีเอ็นเอ เช่น บันทึกว่าเซลล์นี้เคยผ่านสิ่งแวดล้อมที่มีโมเลกุลอาหาร, ยา, สารพิษ ฯลฯ ชนิดนั้นชนิดนี้หรือไม่? มากน้อยแค่ไหน?
งานวิจัยแนวนี้สามารถเอาไปใช้ในการวินิจฉัยโรค เฝ้าระวังสิ่งแวดล้อม หรือศึกษากลไกทางสรีรวิทยาที่เราเข้าถึงได้ยาก เช่น การเอาเซลล์ติดเซ็นเซอร์/ระบบบันทึกแบบนี้ไปใส่ในทางเดินอาหาร ในดิน ในน้ำ เพื่อติดตามการเปลี่ยนระดับโมเลกุลต่างๆ
การบันทึกข้อมูลดิจิทัลลงไปในดีเอ็นเอตรงๆ ยากกว่านั้นเพราะเซ็นเซอร์มีให้เลือกใช้ไม่เยอะ
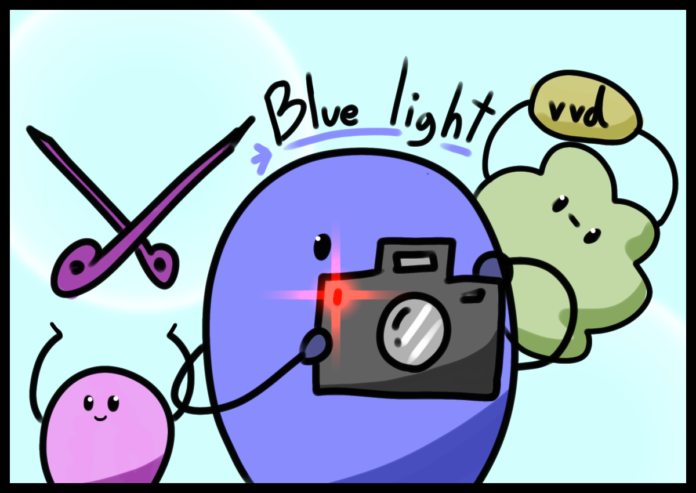
สองปีก่อน (2021) ทีมวิจัยสหรัฐจากมหาวิทยาลัยโคลัมเบีย (Columbia University) บันทึกข้อความตัวอักษรลงแบคทีเรียผ่านสัญญาณไฟฟ้า ในงานนี้ข้อความสั้นๆ อย่าง ” Hello World!” ถูกแปลงเป็นรหัส 0, 1 ที่แทนค่าด้วยการเปิด/ปิดสัญญาณไฟฟ้า
สัญญาณไฟฟ้าเหนี่ยวนำการเปลี่ยนแปลงของไอออนเคมีในอาหารเลี้ยงแบคทีเรียซึ่งถูกวิศวกรรมมาให้รับสัญญาณไอออนพวกนี้โดยเฉพาะและบันทึกลงดีเอ็นเอในรูปแบบการเปลี่ยนแปลงลำดับเบส
ทีมวิจัยสามารถกู้เนื้อหาข้อความนี้กลับมาได้ด้วยการอ่านลำดับเบสที่เปลี่ยนแปลงไปในแบคทีเรีย
Cr : ณฤภรณ์ โสดา
อีกงานของทีมวิจัยสหรัฐจาก MIT เมื่อหกปีก่อน (2017) วิศวกรรมแบคทีเรียให้บันทึกภาพสี ทีมวิจัยติดตั้งระบบเซ็นเซอร์ใหม่ในแบคทีเรียให้ตอบสนองต่อแสงสามช่วงคลื่น (RGB Color) เมื่อฉายภาพลงบนจานเพาะเชื้อแบคทีเรียนี้ แบคทีเรียแต่ละตำแหน่งได้รับแสงแต่ละช่วงคลื่นในสัดส่วนต่างกันขึ้นอยู่กับว่าตำแหน่งนั้นๆ บนภาพมีสีอะไร
แบคทีเรียตอบสองด้วยการผลิตโปรตีนสีออกมาในสัดส่วนตรงกับแสงที่ได้รับไป งานนี้แบคทีเรียยังไม่ได้บันทึกข้อมูลอะไรลงดีเอ็นเอ นอกจากนี้ แบคทีเรียแต่ละตัวบนจานเพาะเชื้อก็ไม่ได้มีข้อมูลเกี่ยวกับตำแหน่งของมัน ดังนั้น ข้อมูลภาพจะสูญสลายไปทันทีที่เราสกัดย้ายแบคทีเรียออกจากตำแหน่งเดิมของมันตอนฉายภาพ
อย่างไรก็ตาม ทีมวิจัยก็ได้แสดงความเป็นไปได้ในการวิศวกรรมเซลล์ให้รับข้อมูลไฟล์ภาพผ่านการฉายแสงโดยตรง
Cr : ณฤภรณ์ โสดา
งานวิจัยล่าสุดของทีมจากมหาวิทยาลัยแห่งชาติสิงคโปร์ (National University of Singapore, NUS) รายงานการบันทึกไฟล์ภาพลงดีเอ็นเอของแบคทีเรียโดยตรง
Chueh Loo Poh หัวหน้าทีมวิจัยจาก NUS ชำนาญเทคโนโลยีการใช้แสงควบคุมการทำงานของยีน (ภาษาทางการเรียกว่า “optogenetics”) เทียบกับการใช้สารเคมีแล้ว “แสง” ต้นทุนถูกกว่าและสามารถปรับความละเอียดได้สูงกว่ามาก
การเปิดปิดยีนด้วยสารเคมีต้องยุ่งยากใช้เวลาในการเติมสารและล้างออก ขณะที่เราเปิดปิดแสงได้ในชั่วพริบตา สารเคมีที่เราให้มักจะแพร่กระจายปนกันมั่วระหว่างเซลล์ที่อยู่ข้างเคียงกัน ขณะที่เราสามารถเล็งเป้าได้แม่นยำว่าเซลล์ตรงตำแหน่งไหนจะโดนหรือไม่โดนแสงคลื่นไหนเมื่อไหร่
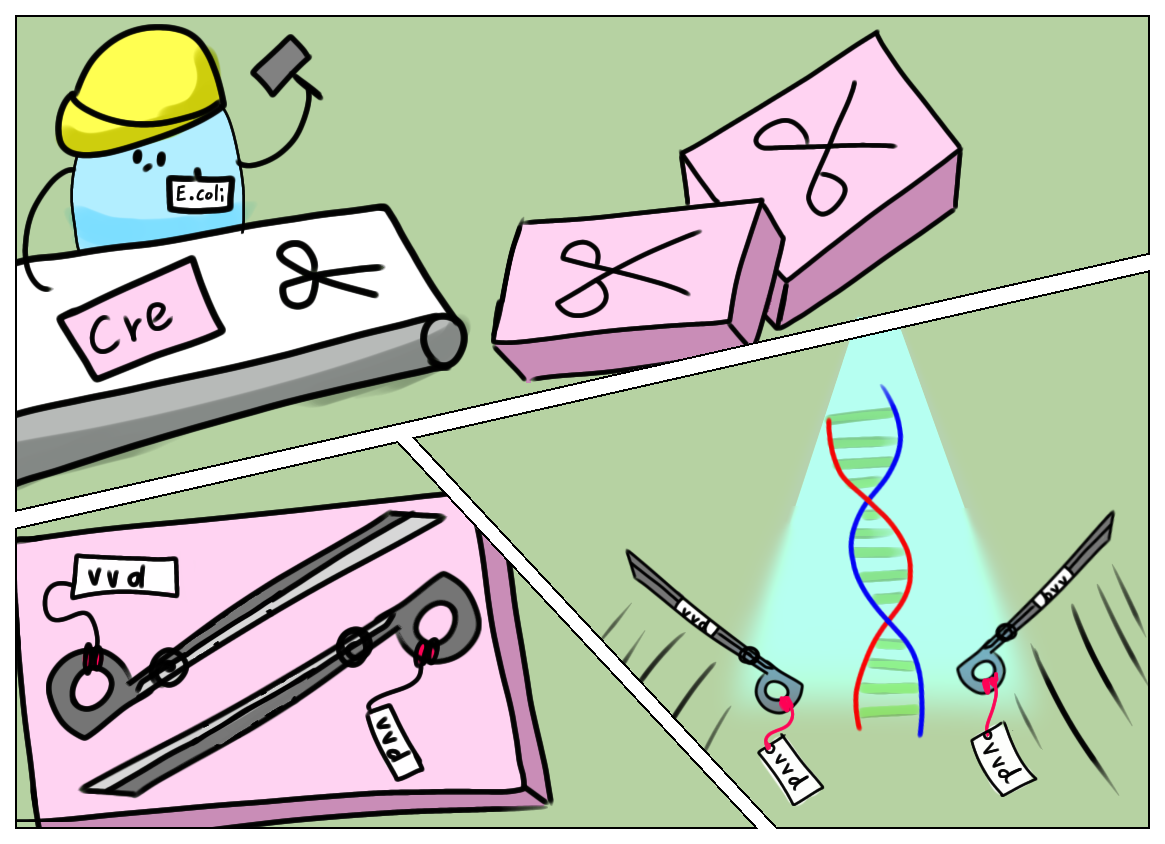
ทีมของ Poh วิศวกรรมแบคทีเรีย E.coli ให้ผลิตเอนไซม์ชื่อ Cre ทำหน้าที่เหมือนกรรไกรตัดทำเครื่องหมายบนดีเอ็นเอ ณ ตำแหน่งจำเพาะในเซลล์แทนการบันทึกข้อมูล
กรรไกรนี้ถูกออกแบบให้แยกเป็นสองส่วน ยังไม่สามารถตัดดีเอ็นเอได้จนกว่าจะมาประกอบร่างกันเป็นกรรไกรที่สมบูรณ์
กรรไกรแต่ละส่วนพ่วงอยู่กับชิ้นส่วนของโปรตีนอีกตัวชื่อ Vvd ยืมมาจากราชนิด Neurospora crassa ตามธรรมชาติราชนิดนี้ใช้ Vvd เป็นเซ็นเซอร์รับแสงสีฟ้าสำหรับตอบสนองของรอบเวลากลางวัน/กลางคืน
ชิ้นส่วน Vvd จะมาประกอบร่างกันเมื่อได้รับแสงสีฟ้าและพาชิ้นส่วนกรรไกร Cre ที่พวกอยู่มาด้วย ทั้งระบบนี้ทำให้ทีมของ Poh ได้ E.coli ที่บันทึกข้อมูลว่าเคยได้/ไม่ได้รับแสงสีฟ้ามาก่อน
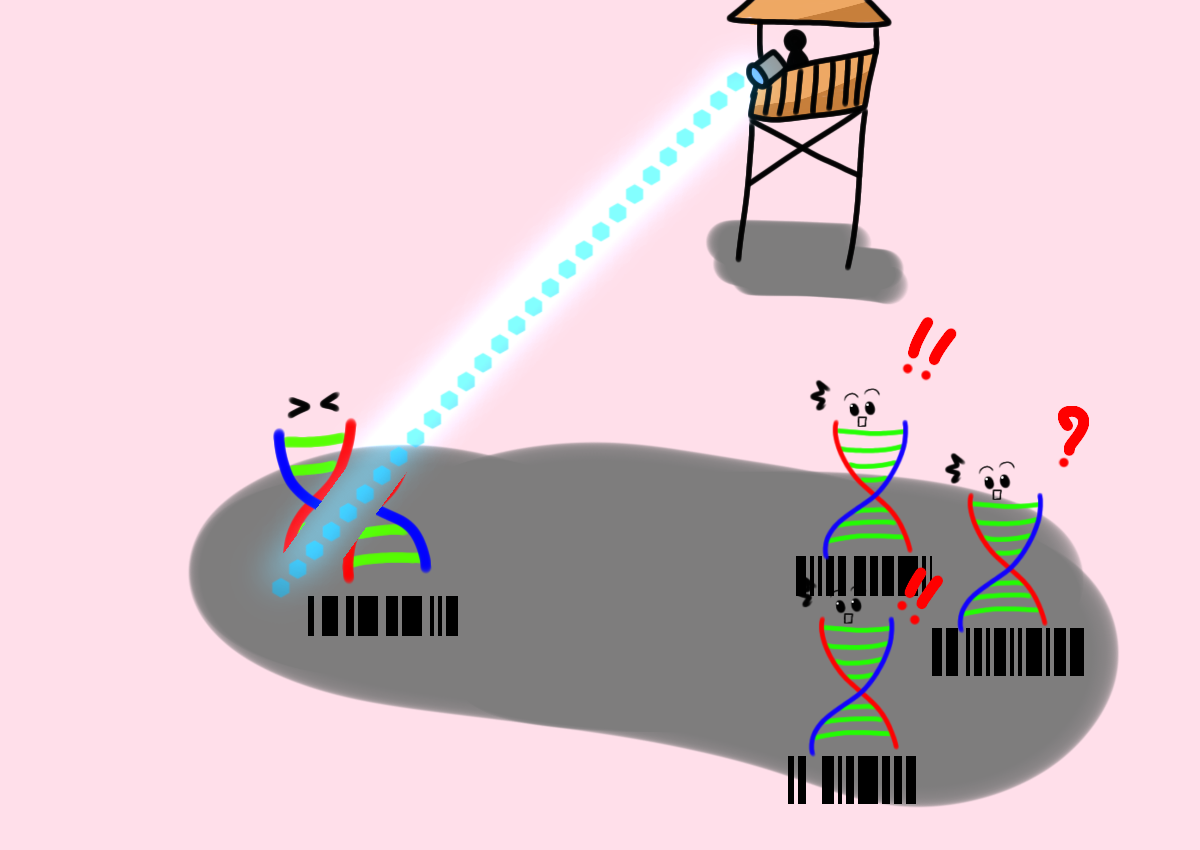
ข้างตำแหน่งเป้าหมายการตัดของ Cre ทีมวิจัยได้ติดตั้งลำดับเบสดีเอ็นเอเพิ่มเติมมาเพื่อเป็นบาร์โค้ดระบุตำแหน่งเซลล์
ทีมวิจัยเลี้ยงเซลล์บนถาดเพาะเชื้อขนาด 12 x 8 หลุม เซลล์แต่ละหลุมมีดีเอ็นเอบาร์โค้ดต่างกันไป
ทีมวิจัยฉายแสงสีฟ้าลงบนจานเพาะเป็นรูปหรือตัวอักษรต่างๆ แต่ละหลุมแทนหนึ่งพิกเซล (pixel) ของภาพนั้นๆ
ถ้าหลุมนั้นได้รับแสงดีเอ็นเอของเซลล์ในหลุมก็จะถูกตัดเป็นรอยบันทึกไว้แต่ถ้าไม่ได้รับแสงดีเอ็นเอก็จะคงสภาพเดิม
ทีมวิจัยสามารถเอาเซลล์จากทุกหลุมมาเทรวมกันเก็บในหลอดหลอดเดียว
เวลาจะดึงข้อมูลภาพกลับมาก็ใช้การอ่านลำดับเบสของดีเอ็นเอจากแบคทีเรียที่เก็บไว้
ลำดับเบสที่อ่านได้บอกบาร์โค้ดและร่องรอยการตัดดีเอ็นเอซึ่งสามารถแปลงกลับมาเป็นข้อมูลว่าพิเซลไหนเคยหรือไม่เคยได้รับแสง
ระบบบาร์โค้ดระบุตำแหน่งนี้เรียบง่ายแต่ว่าชาญฉลาดมาก ไม่เพียงแต่ทีมวิจัยสามารถจะเก็บข้อมูลพิเซลของภาพในหลอดเดียวเท่านั้น แต่ยังสามารถเก็บข้อมูลจากภาพหลายๆ ภาพรวมกันในหลอดเดียวอีกด้วย
การเพิ่มบาร์โค้ดอีกชิ้นที่สามารถระบุว่าดีเอ็นเอชิ้นดังกล่าวมาจากภาพไหน ทีมวิจัยใช้เทคนิคการเพิ่มจำนวนดีเอ็นเอจำเพาะตำแหน่งอย่าง PCR ในการดึงเอาข้อมูลเฉพาะรูปเอามาวิเคราะห์ได้โดยไม่จำเป็นต้องอ่านลำดับเบสทั้งหมด
ระบบนี้ทำหน้าที่เหมือนกับ RAM (Random Access Memory) ในเครื่องคอมพิวเตอร์ปกติ วิธีนี้ใช้การได้แม้แต่กับตัวอย่างดีเอ็นเอที่ผ่านการเจือจางมาแล้วถึงห้าหมื่นเท่า
ทีมวิจัยทดสอบความทนถาวรของระบบบันทึกนี้ ด้วยการเอาดีเอ็นเอที่เก็บรวมกันไปแช่แข็งแช่แข็ง ผ่านความร้อน 60 องศาเซลเซียส หรือไว้ที่อุณหภูมิห้องได้อย่างน้อยหนึ่งสัปดาห์ และสามารถเอาไปผ่านรังสีอัลตราไวโอเลตนานอย่างน้อยหนึ่งชั่วโมงโดยข้อมูลที่ได้เก็บไว้ไม่ได้เสื่อมสลายไป
ทีมวิจัยได้ยังทดลองบันทึกข้อมูลภาพมากกว่าหนึ่งภาพลงบนถาดเพาะแบคทีเรียชุดเดียวกันโดยใช้แสงต่างช่วงคลื่น (สีฟ้าและสีแดง) และสร้างระบบตรวจจับและระบบการเขียนดีเอ็นเอที่ตอบสนองต่อแสงทั้งสองช่วงคลื่นนี้แยกกัน
แม้ว่างานวิจัยนี้เป็นการทดสอบเบื้องต้นบนถาดเลี้ยงเซลล์จึงได้ภาพที่มีความละเอียดต่ำแค่ไม่ถึงร้อยพิกเซล
แต่ก็ไม่ยากที่เราจะเพิ่มความละเอียดให้สูงขึ้นด้วยการทำหลุมให้เล็กลงหรือใช้ตัวปรินต์ความละเอียดสูงเนื่องจากระบบนี้ไม่ต้องการปริมาณดีเอ็นเอและจำนวนเซลล์ในการบันทึกข้อมูลไม่มากนัก นอกจากนี้ เราอาจจะเอาข้อมูลดิจิทัลอื่นๆ เช่น QR code มาบันทึกอยู่ในรูปแบบข้อมูลพิกเซลสองหรือสามมิติด้วยวิธีนี้
ความสามารถในการย้ายถ่ายโอนข้อมูลไปมาระหว่าง “โลกดิจิทัล” กับ “โลกชีววิทยา” ทำให้เราสามารถจะใช้ประโยชน์จากทั้งสองโลกนี้ ทั้งการสร้าง ประมวล และวิเคราะห์ข้อมูลความละเอียดสูงที่ต้องใช้พลังการคำนวณมหาศาลแบบโลกดิจิทัล และระบบการทำงานแบบโลกชีววิทยาที่แม้จะช้ากว่าแต่ก็ยั่งยืนกว่าทั้งในแง่ความทนทานของข้อมูล การประหยัดพลังงาน เป็นมิตรกับสิ่งแวดล้อม
แถมยังประสานงานอย่างลงตัวกับกลไกตามธรรมชาติในเซลล์ของเรา
Cr : ณฤภรณ์ โสดา
สะดวก ฉับไว คุ้มค่า สมัครสมาชิกนิตยสารมติชนสุดสัปดาห์ได้ที่นี่https://t.co/KYFMEpsHWj
— MatichonWeekly มติชนสุดสัปดาห์ (@matichonweekly) July 27, 2022